組織について/育種研究センター(Research Center for Aquatic Breeding)
ゲノム育種グループ(Genomics Breeding Group)
ゲノム育種グループは、基本的な育種技術や形質評価技術及び系統の作出と安全性評価等に関する研究開発を行っています。
主にブリ、ヒラメ、トラフグなどの海産魚を対象に、病気に強い、成長が速いなどといった優良な形質の評価法の開発、それらの形質を持つ家系を選抜育種するための指標となる遺伝子マーカーの開発、選抜に頼らない人為的な変異導入法などの開発に加え、実際に選抜系統を作出してその生物特性や有用性の評価に取り組んでいます。また、主にサケ・マス類、コイなどの淡水魚を対象に、遺伝子組換え魚や外来種による遺伝資源のかく乱の防止や天然資源の生物多様性の保全の観点から、遺伝子組換え魚類の検出や生物多様性への影響評価のための研究開発に取り組んでいます。
研究メンバー
グループ長 岡本 裕之 主任研究員 正岡 哲治 主任研究員 尾崎 照遵 研究員(任期付) 内野 翼
研究について
海産魚(ブリ)の育種研究(図1)
―ハダムシの付きにくいブリの作出―
ブリ類は日本で最も多く養殖されている魚種ですが、天然の稚魚を捕獲してそれを養殖するという手法をとっているために、毎年多くの感染症の被害を受けます。特に、ハダムシがブリの表皮について炎症を起こし、ブリがハダムシを落とすために体を網にこすりつけ2次感染を起こして大量に死ぬことがあります。そのため、ハダムシの寄生を避けるためにハダムシの卵が付着した網を代える必要があり、多くの労力と人手が必要となります。そこで、ハダムシが付きにくいブリの家系を作るための研究を行っています。

図をクリックすると拡大 |
―ハダムシの付きにくいブリの作出―(図1)
天然から捕ってきたブリにハダムシの感染実験を行い、ハダムシの付きにくいブリを見つけて交配をしたブリの家系の中で、共通して持つ染色体の場所(遺伝子座)を特定して、さらに、その家系のブリを選別して交配を行い、その稚魚の感染実験を行っているところです。 |
海産魚(ヒラメ)の育種研究(図2)
―細菌感染症抵抗性のヒラメの育種―
ヒラメは、我が国では多くの場合、陸上の施設で養殖されています。このような閉鎖的な飼育環境では、他の養殖対象種に較べてレンサ球菌やエドワジエラなどの細菌感染やリンホシスチス等のウイルスに感染すると蔓延しやすく、毎年多くの病気による被害が起こっています。グループでは、遺伝子の情報を利用して、レンサ球菌感染症に抵抗性を持つヒラメの家系を作ることに挑戦しました。現在は生産現場での実証試験により実用化に必要な問題点の抽出を行っています。今後は、被害の大きいエドワジエラ症に対する抵抗性についても調べます。
DNA情報を用いた種判別の応用(図3)
―貝類(アコヤガイ)の育種―
アコヤガイと近縁種は、殻の形態にいろいろな変異があることや、幼生や稚貝の形態はよく似ているため、貝殻の形態等で種を判別するのは困難です。そこで、DNAの違いを利用して、アコヤガイと近縁種を判別する手法を開発しました。
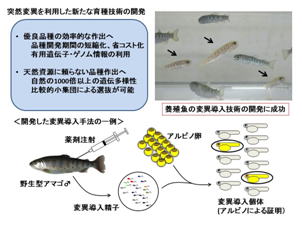
新規育種手法の開発研究(図4)
―養殖魚の突然変異育種とゲノム編集―
農業や畜産で数千年かけて行われてきた育種は、これからの養殖業においても、産業の発展、食の安定供給を図る上で重要な研究テーマの一つと考えられます。育種を進める最大の問題は、開発時間の長さです。また、多くの個体(魚)を飼育するためのスペース、コスト、労力も無視できない大きな問題です。こうした問題を軽減し効率化する一つの技術として、「突然変異を利用した育種技術の開発」を行っています。育種の主要な考え方の一つとして、様々な遺伝的形質(遺伝的多様性)を持つ集団の中から我々にとって、有用な形質を持つ個体を選抜していくことがあげられますが、本研究では、多くの天然集団から選抜するのではなく、突然変異を利用して小さな集団に多くの遺伝的多様性を持たせ、これを選抜する集団とすることによって、育種にかかる時間、コスト等を低減し、効率的に品種を作出していく技術の開発を目指しています。
また新規育種技術としてゲノム編集技術の利用についても検討していきます。
外来魚の駆除・根絶技術の開発(図5)
―新たな外来魚の根絶技術の考案―
侵略的外来魚(ブルーギル、オオクチバスなど)は、在来希少種や固有の生態系の破壊などの要因の一つとして考えられています。これらの魚類を駆除するため、物理的な捕獲(網、釣り、電気ショック等)、池干しおよび産卵巣の破壊などが続けられていますが、取り残しなく駆除することは大変困難です。そこで、物理的捕獲に加え、不妊化魚を使って駆除・根絶するという新たな手法を考案し、必要な技術開発を進めます。
研究課題
- 遺伝子制圧技術による外来魚の根絶のための実証魚の開発(2017-2019)
- 細菌性疾病抵抗性ヒラメの実用化実証・対策研究(2018-2019)
- ブリの性決定・耐病性・成長に関わる遺伝子の機能解析魚の作出(2018-2019)
- 高成長ブリ類家系を天然魚から短期間で作出可能な新たな育種技術開発 (2016-2020:農林水産技術会議競争的資金)
- ハダムシ抵抗性に関与する遺伝子座の同定と選抜育種用のマーカーの開発 (2014-2018:農林水産技術会議プロジェクト研究)
- カンパチのゲノム解析と遺伝子地図の作成 (2014-2018:農林水産技術会議プロジェクト研究)
- 水産有用種の育種の高速化のためのゲノミックセレクションに関する日米国際共同研究 (2016-2018:交付金 (国際共同))
発表論文
- 正岡哲治,名古屋博之,岡本裕之,荒木和男,藤原篤志,小林敬典 (2018)屋外大型飼育池におけるコイとフナ類の自然交雑の確認.DNA多型 26, 25-29
- Masaoka T, Okamoto H, Araki K, Nagoya H, Fujiwara A, Kobayashi T (2018) Distinction between non-native and Japanese native cyprinid fishes based on mitochondrial 16S ribosomal RNA gene regions. DNA Testing 10, 31-41
- 正岡哲治,名古屋博之,岡本裕之,荒木和男 (2018) X線照射による遺伝子組換えアマゴの不妊化.水産育種 48,117-123
- 尾崎 照遵,荒木 和男,青木 純哉,嶋田 幸典,岡本 裕之,薄 浩則,奥澤 公一,吉田 一範,野田 勉 (2018) Paradigm shift in fish breeding; marker-assisted selection to genomic selection. Proceedings of the 44th U.S.-Japan Aquaculture Panel Symposium. UJNR Aquaculture Panel Symposium 20-24
- Andrew Severin,Catherine Purcell,Mark Drawbridge,Rick Masonbrink,Arun Seetharam,尾崎 照遵,荒木 和男,薄 浩則,奥澤 公一,乙竹 充,伊藤 文成,John Hyde (2018) Seriola genomics and the knowledge repository Serioladb.org. Proceedings of the 44nd U.S.-Japan Aquaculture Panel Symposium. UJNR Aquaculture Panel Symposium 52- 57
- 尾崎 照遵 (2017) 遺伝学の知識を生かして優良魚の作出確率を上げる飼育手法 水産育種 47(1), 47-48
- Masaoka T, Okamoto H, Araki K, Nagoya H, Fujiwara A, Kobayashi T (2017) Distinction of unknown Pinctada species based on 28S and 16S ribosomal RNA gene regions and IGS regions of nuclear ribosomal RNA genes. DNA Testing 9, 23-34
- Mutagenesis and Genome Editing for Aquaculture Fish Species: Modification of SCPP Genes in Tiger Pufferfish (Takifugu rubripes)(2017)岡本裕之、石川卓、甲斐渉、西木一生、岩崎裕貴、岸本謙太、村上悠、木下政人、藤原篤志、Genetics and Breeding in Aquaculture. Proceedings of the 44th U.S.-Japan Aquaculture Panel Symposium、NMFS-F巻/SPO-175号、26-32
- キンギョを用いた突然変異誘発(2017)岡本裕之、黒田拓男、澤田知希、白木谷卓哉、金田康見、荒川純平、尾崎照遵、石川卓、宮本淳司、岡村康弘、水産育種、47巻/1号、49-54
- Development and characterization of 24 polymorphic microsatellite markers for bluegill sunfish, Lepomis macrochirus(2017)河村功一、藤原篤志、山田充哉、古川航、栗田潤、岡本裕之、 Genes Genomics、DOI 10.1007/s13258-017-0531-x
- 正岡哲治,名古屋博之,岡本裕之,荒木和男,藤原篤志,小林敬典 (2016) PCRを利用したコイとフナ類の雑種判別.DNA多型 24, 23-26
- Kanonkporn Kessuwan, 久保田諭, Qi Liu, 佐野元彦, 岡本信明, 坂本崇, 山下浩史, 中村洋路, 尾崎照遵 (2016) Detection of Growth-Related Quantitative Trait Lociand High-Resolution Genetic Linkage Maps Using Simple Sequence Repeat Markers in the Kelp Grouper.(Epinephelus bruneus). Marine Biotechnology 18, 57-84
- Masaoka T, Okamoto H, Araki K, Nagoya H, Fujiwara A, Kobayashi T (2016) Distinction between Pinctada species based on nuclear and mitochondrial ribosomal RNA gene regions. DNA Testing 8, 9-21
- Aoki J, KaiW, Kawabata Y, OzakiA, Yoshida K, Koyama T, Sakamoto T, Araki K (2015) Second generation physical and linkage maps of yellowtail (Seriola quinqueradiata) and comparison of synteny with four model fish GMC Genomics 16(406) 2-11
- Koyama T, Ozaki A, Yoshida K, Suzuki J, Fuji K, Aoki J, Kai W, Kawabata Y, Tsuzaki T, Araki K, Sakamoto T (2015) Identification of Sex-Linked SNPs and Sex-Determination Regions in the Yellowtail Genome. Marine Biotechnology 17(4), 502-510
- Masaoka T, Oku H, Okamoto H, Arak, K, Nagoya H, Yanagimoto T, Fujiwara A, Kobayashi T (2015) Distinction of hybrids between Salvelinus leucomaenis and Salmo salar using aromatase gene. DNA Testing 7, 27-35
- Aoki J, Kai W, Ozaki A, Thsuzaki T, Fuji K, Koyama T, Sakamoto T, Araki K (2014) Construction of a radiation hybrid panel and the first yellow tail (Seriola quinqueradiata) radiation hybrid map using a nanofluidic dynamic array. BMC Genomics 15(165), 1-10
- 藤加菜子, 小山喬, 久保田諭, 吉田 一範, 尾崎 照遵, 津崎 龍雄, 甲斐渉, 青木純哉, 小山喬, 中川雅弘, 堀田卓朗, 津崎龍雄,荒木和男荒木 和男, 坂本崇, 岡本信明 (2014) Construction of a high-coverage bacterial artificial chromosome library and comprehensive genetic linkage map of yellowtail Seriola quinqueradiata. BMC research note, 7-200, 1-10
- 正岡哲治,名古屋博之,岡本裕之,荒木和男,藤原篤志,小林敬典 (2014) DNAマーカーによるコイとフナ類の雑種判別.DNA多型 22, 80-83
- Ozaki A, Yoshida K, Fuji K, Kubota S, Kai W, Aoki J, Kawabata Y, Suzuki J, Akita K, Koyama T, Nakagawa M, Hotta T, Tsuzaki T, Araki K, Sakamoto T (2013) Quantitative Trait Loci (QTL) Associated with Resistance to a Monogenean Parasite (Benedenia seriolae) in Yellowtail (Seriola quinqueradiata) through Genome Wide Analysis. PLOS ONE, 8(6), e64987, 1-14
- Qi Liu, 坂本崇, 岡本信明, 山下浩史, 高木基裕, 重信裕弥, 菅谷琢磨, 中村洋路, 佐野元彦, Wuthisuthimethavee S, 尾崎照遵, (2013) A genetic linkage map of kelp grouper (Epinephelus bruneus) based on microsatellite markers. Aquaculture 414–415, 63–81
- Masaoka T, Samata T, Nogawa C, Baba H, Aoki H, Kotaki T, Nakagawa A, Sato M, Fujiwara A, Kobayashi T (2013) Shell matrix protein genes derived from donor expressed in pearl sac of Akoya pearl oyster (Pinctada fucata) under pearl culture. Aquaculture 384-387, 56-65
- Masaoka T, Okamoto H, Araki K, Nagoya H, Fujiwara A, Kobayashi T (2012) Identification of the hybrid between Oryzias latipes and Oryzias curvinotus using nuclear gene and mitochondrial gene region. Marine genomics 7, 37-41
- Masaoka T, Okamoto H, Araki K, Nagoya H, Fujiwara A, Kobayashi T (2012) Sterility of triploid F2 hybrid between Oryzias latipes and Oryzias curvinotus. Fish genetics and breeding science 41, 43-49
〔特許〕
- 知財管理番号:245 特願2013-55979 公開番号:特開2014-180233 「ブリ類の性識別方法」ブリ類の性を識別するための方法を提供する。審査請求期限日:2016/3/19
- 知財管理番号:294 特願2017-42793 「ブリ類のハダムシ抵抗性検査方法」ブリ類のハダムシ抵抗性を検査するための方法 審査請求期限日:2020/3/7
- 知財管理番号:295 特願2016-132224 「成長性遺伝形質を有するアカマダラハタの識別方法(権利放棄)」アカマダラハタの高成長性マーカーを開発して、個体を殺すことなく高成長性の形質を持つ魚を判別することのできる方法を提供する。
- 「突然変異養殖魚」:特許 第5849305号、2015年12月11日 岡本裕之
- 「ブリ類の性識別方法」:PCT/JP 2013-055979、2013年2月8日 坂本崇、小山喬、尾崎照遵、荒木和男、吉田一範、津崎龍雄